文書の過去の版を表示しています。
目次
Rによる生存時間解析
データの書き方
多くのRの解析プログラム(パッケージ)では、データフレームという型のオブジェクトでデータを与えることができる。
| # | 時間 | 故障/打ち切り |
| 1 | 1.5 | 故障 |
| 2 | 5.3 | 故障 |
| 3 | 2.5 | 故障 |
| 4 | 7.8 | 故障 |
| 5 | 6.2 | 故障 |
| 6 | 4.1 | 故障 |
| 7 | 8.2 | 故障 |
| 8 | 10.3 | 故障 |
| 9 | 1.5 | 故障 |
| 10 | 13.4 | 故障 |
上のデータをRに入力するには、次のコードを用いる。
data.frame(time=c(1.5,5.3,2.5,7.8,6.2,4.1,8.2,10.3,1.5,13.4),
status=c(1,1,1,1,1,1,1,1,1,1))
これを実行すると次のように表示される。
time status 1 1.5 1 2 5.3 1 3 2.5 1 4 7.8 1 5 6.2 1 6 4.1 1 7 8.2 1 8 10.3 1 9 1.5 1 10 13.4 1
打ち切りデータもある場合には、次のように打ち切りに対応するstatusを0にする。
| # | 時間 | 故障/打ち切り |
| 1 | 1.5 | 故障 |
| 2 | 5.3 | 故障 |
| 3 | 2.5 | 故障 |
| 4 | 7.8 | 故障 |
| 5 | 6.2 | 故障 |
| 6 | 4.1 | 故障 |
| 7 | 8.2 | 故障 |
| 8 | 10.0 | 打ち切り |
| 9 | 1.5 | 故障 |
| 10 | 10.0 | 打ち切り |
data.frame(time=c(1.5,5.3,2.5,7.8,6.2,4.1,8.2,10.0,1.5,10.0),
status=c(1,1,1,1,1,1,1,0,1,0))
time status 1 1.5 1 2 5.3 1 3 2.5 1 4 7.8 1 5 6.2 1 6 4.1 1 7 8.2 1 8 10.0 0 9 1.5 1 10 10.0 0
データの代入
オブジェクト(変数)へのデータの代入には、代入演算子「=」を用いる。
X = data.frame(time=c(1.5,5.3,2.5,7.8,6.2,4.1,8.2,10.3,1.5,13.4),
status=c(1,1,1,1,1,1,1,1,1,1))
これで、先ほどのデータは「X」というオブジェクトの中に含まれる。
時間timeだけを取り出すには「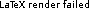 time
</code>
故障か打ち切りかを表すメンバstatusを取り出すのも同様である。
time
</code>
故障か打ち切りかを表すメンバstatusを取り出すのも同様である。
X$status
survivalパッケージの読み込み
生存時間解析のためのパッケージを読み込んでおく。
library(survival)
これを実行しておかないと、すぐ次のコマンドの実行時に赤い文字でエラーが表示される。
生存曲線と故障率曲線のノンパラメトリック推定
特定の確率分布を仮定せずに母数や分布を推定する方法を、ノンパラメトリックな推定法と呼ぶ。
カプランさんとマイヤーさんが提案した、生存関数をノンパラメトリックに推定する方法がカプラン・マイヤー法である。カプラン・マイヤー推定量とも呼ばれる。生存時間データから寿命分布を推定する際に、まずは何も仮定せずに生存関数を推定して眺めてみることが、第一歩となる。
survfit(Surv(time, status)~1,data=X)
まず表示されるのはサンプル数n、イベント数events、寿命のメディアン、寿命のメディアンの95%下側信頼区間、寿命のメディアンの95%上側信頼区間、である。
Call: survfit(formula = Surv(time, status) ~ 1, data = X)
n events median 0.95LCL 0.95UCL
10.00 10.00 5.75 2.50 NA
関数survfitは、生存関数をカプラン・マイヤー法で推定してくれる。他にCoxの比例ハザードモデルを推定した結果や、加速寿命時間モデルを推定した結果からも、生存関数を推定してくれるが、これらはこの説明の想定を超えるので、これ以上は述べない。
カプラン・マイヤー推定量で生存関数を推定した結果を、オブジェクトX.survfitに保存する。
X.survfit <- survfit(Surv(time, status)~1,data=X)
推定結果は、関数summaryを用いて表示できる。
summary(X.survfit)
これで時点ごとの生存関数の推定値や、標準誤差、信頼区間が表示される。
Call: survfit(formula = Surv(time, status) ~ 1, data = X) time n.risk n.event survival std.err lower 95% CI upper 95% CI 1.5 10 2 0.8 0.1265 0.5868 1.000 2.5 8 1 0.7 0.1449 0.4665 1.000 4.1 7 1 0.6 0.1549 0.3617 0.995 5.3 6 1 0.5 0.1581 0.2690 0.929 6.2 5 1 0.4 0.1549 0.1872 0.855 7.8 4 1 0.3 0.1449 0.1164 0.773 8.2 3 1 0.2 0.1265 0.0579 0.691 10.3 2 1 0.1 0.0949 0.0156 0.642 13.4 1 1 0.0 NaN NA NA
これを関数plotを用いて表示することができる。
plot(X.survfit)
legend("topright",lty=c(1,2),legend=c("Kaplan-Meier Est.", "95% Conf. Int."),cex=0.5)
二行目は凡例を追加するコマンドである。
実線の折れ線が、カプラン・マイヤー法で推定した生存関数である。1から生存関数を引けば、累積分布関数の推定ができる。このグラフからまずは、寿命分布の密度関数が一山か、二山以上か、を読み取ろうとする。 survfitに収められている、各時点でのリスクセットの大きさn.riskとその時点での故障数n.eventの比を計算すると、故障率曲線も推定できる。
X.survfit$n.event/X.survfit$n.risk
これを縦軸に、時間を横軸にとったグラフ
plot(X.survfit$time,X.survfit$n.event/X.survfit$n.risk,
xlab="Time",ylab="Failure Rate")
が故障率曲線のノンパラメトリックな推定値となる。
ノンパラメトリックな推定量の長所と短所
ノンパラメトリックな推定量は、確率分布を仮定しないので、当初から色眼鏡で見ることがなく、データをそのまま吟味できるという長所を持つ。実際に観測された時点から先の予測ができない、という短所も合わせ持つ。また、精度よく確率分布を推定するには、とても大きいサンプルを必要とすることも、短所である。
信頼性は時間に関する品質である。販売あるいは投入したすべてのシステムが故障した後に、寿命分布を正しく推定できても、意味がない。可能ならば市場に投入したり現場で運用を開始する前や大量生産に踏み切る前の、設計開発段階や試作段階で、寿命分布を推定しておきたい。そうして、品質保証制度に基づく補償請求に応じる費用や、運用期間中の総保守費用を予測しておきたい。更に可能ならば、それらの費用を削減するように、設計や開発を行いたいのである。
そこでシステムや製品の信頼性や保全性を設計・計画する際には、具体的に寿命が従う確率分布(寿命分布)をパラメトリックに推定して、それに基づいて対応することが通例となっている。
パラメトリックな寿命分布の例
ワイブル分布
対数正規分布
ガンマ分布
グンベル分布
故障時間データからのパラメトリックな寿命分布の推定法
すべてが故障しているデータ
すべてが故障しているデータの場合は、MASSパッケージの中の関数fitdistrを用いて、確率分布の未知母数の最尤推定値と標準誤差を得ることができる。まず、MASSパッケージを読み込む。
library(MASS)
次に、関数fitdistrを用いて、ワイブル分布を仮定し、その2つの母数を推定してみる。
fitdistr(X$time, densfun="weibull")
その結果、次の数値を得る。
shape scale 1.6822280 6.8219220 (0.4279334) (1.3515357)
ワイブル分布には形状母数shapeと尺度母数scaleがある。それぞれの点推定値は1.6822280と6.8219220であり、標準誤差は括弧の中に表示される。
これらは、次のようにしても表示される。
X.fit.weibull <- fitdistr(X$time, densfun="weibull") names(X.fit.weibull) X.fit.weibull$estimate X.fit.weibull$sd X.fit.weibull$loglik
最後の数値は、最尤推定法で最大化した対数尤度関数の最大値である。この値を参考に、寿命分布を選ぶこともある。
対数正規分布も推定しておく。
X.fit.lognormal <- fitdistr(X$time, densfun="lognormal") names(X.fit.lognormal) X.fit.lognormal$estimate X.fit.lognormal$sd X.fit.lognormal$loglik
同様に、ガンマ分布。
X.fit.gamma <- fitdistr(X$time, densfun="gamma") names(X.fit.gamma) X.fit.gamma$estimate X.fit.gamma$sd X.fit.gamma$loglik
関数
完全データについて、これら三つの確率分布を推定し、ヒストグラムと比較するための関数を用意した。
draw.histgram.and.densities <- function(X,hist=TRUE) {
require(MASS)
est.weibull <- fitdistr(X$time,densfun="weibull")
est.lnorm <- fitdistr(X$time,densfun="lognormal")
est.gamma <- fitdistr(X$time,densfun="gamma")
if(hist==TRUE) {
hist(X$time,freq=FALSE,ylim=c(0,0.3),
xlab="Lifetime")
plot(function(x){return(dlnorm(x,meanlog=est.lnorm$estimate[1],
sdlog=est.lnorm$estimate[2]))},
xlim=c(0,12),
col="blue",
add=TRUE)
} else {
plot(function(x){return(dlnorm(x,meanlog=est.lnorm$estimate[1],
sdlog=est.lnorm$estimate[2]))},
xlim=c(0,12),
col="blue",xlab="Lifetime",ylab="Density")
}
plot(function(x){return(dweibull(x,shape=est.weibull$estimate[1],
scale=est.weibull$estimate[2]))},
xlim=c(0,12),
col="red",
add=TRUE)
plot(function(x){return(dgamma(x,shape=est.gamma$estimate[1],
rate=est.gamma$estimate[2]))},
xlim=c(0,12),
col="green",
add=TRUE)
legend("topright",
col=c("blue","red","green"),
lty=c(1,1,1),
legend=c("lognormal","weibull","gamma"))
return(list(weibull=est.weibull,lnorm=est.lnorm,gamma=est.gamma))
}
オプションのhistをhist=FALSEとすると、ヒストグラムは描かず、密度関数のみを描くようになる。
またカプラン・マイヤー法で推定した生存関数と共に描くための関数も用意した。
compare.KM.and.distributions <- function(fit,xlim=NULL) {
require(survival)
X.km <- survfit(Surv(time,status)~1,data=X)
if(!is.null(xlim)) {
plot(X.km,
xlab="Time",
ylab="Survival Function",xlim=xlim)
plot(function(x) pweibull(x,shape=X.fits$weibull$estimate[1],scale=X.fits$weibull$estimate[2],
lower.tail=FALSE),
xlim=xlim,add=TRUE,col="green")
plot(function(x) plnorm(x,meanlog=X.fits$lnorm$estimate[1],sdlog=X.fits$lnorm$estimate[2],
lower.tail=FALSE),
xlim=xlim,add=TRUE,col="blue")
plot(function(x) pgamma(x,shape=X.fits$gamma$estimate[1],rate=X.fits$gamma$estimate[2],
lower.tail=FALSE),
xlim=xlim,add=TRUE,col="orange")
} else {
plot(X.km,
xlab="Time",
ylab="Survival Function",xlim=xlim)
plot(function(x) pweibull(x,shape=X.fits$weibull$estimate[1],scale=X.fits$weibull$estimate[2],
lower.tail=FALSE),
xlim=c(0,max(X.km$time)),add=TRUE,col="green")
plot(function(x) plnorm(x,meanlog=X.fits$lnorm$estimate[1],sdlog=X.fits$lnorm$estimate[2],
lower.tail=FALSE),
xlim=c(0,max(X.km$time)),add=TRUE,col="blue")
plot(function(x) pgamma(x,shape=X.fits$gamma$estimate[1],rate=X.fits$gamma$estimate[2],
lower.tail=FALSE),
xlim=c(0,max(X.km$time)),add=TRUE,col="orange")
}
legend("topright",lty=c(1,2,1,1,1),col=c("black","black","green","blue","orange"),
legend=c("Estimate","95% Confidence Interval",
paste("Weibull",round(X.fits$weibull$loglik,5)),
paste("Lognormal",round(X.fits$lnorm$loglik,5)),
paste("Gamma",round(X.fits$gamma$loglik,5))),
cex=0.5)
}
これで、どの分布に最も近いかを検討できる。
一部が打ち切られているデータ
一部の寿命の観測が、故障まで継続されず、途中で打ち切られているデータを、打ち切りデータという。打ち切りデータの場合は、MASSパッケージの中の関数fitdistrは用いることができない。代わりに、survivalパッケージの中の関数survregを用いる。この関数も確率分布の未知母数の最尤推定値と標準誤差を得ることができる。まずsurvivalパッケージを読み込む。
library(survival)
